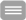Beteiligung von S100A11 an der Reparatur von DNA-Doppelstrangbrüchen
S100-Proteine können durch Bindung an Interaktionspartner deren Aktivität modulieren. Sie sind in verschiedene zelluläre Prozesse, beispielsweise die Zellzyklus-Regulation, Zellwachstum und Zellmotilität involviert. Wir konnten eine funktionelle Kolokalisierung von S100A11 mit Proteinen detektieren, die an der homologen Rekombinationsreparatur von DNA-Doppelstrangbrüchen (DSB) beteiligt sind. Hierbei hängt das Erkennen der Orte mit DSB durch RAD54B von dessen Interaktion mit S100A11 ab. Im Rahmen dieses Projekts wollen wir detailliert untersuchen, wie S100A11 in den DNA-Reparaturprozess eingebunden ist.
Beteiligte Wissenschaftler
Fördernde Institutionen
- Wilhelm-Sander-Stiftung
Ausgewählte projektbezogene Publikationen
- Murzik U., Hemmerich P., Weidtkamp-Peters S., Ulbricht T., Bussen W., Hentschel J., von Eggeling F., Melle C.:
Rad54B targeting to DNA double-strand break repair sites requires complex formation with S100A11.
Mol. Biol. Cell 19, 2926-2935 (2008). - Gorsler T., Murzik U., Ulbricht T., Henschel J., Hemmerich P., Melle C.:
DNA damage-induced translocation of S100A11 into the nucleus regulates cell proliferation.
BMC Cell Biol. 11, 100 (2010). - Foertsch F., Teichmann N., Kob R., Hentschel J., Laubscher U., Melle C.:
S100A11 is involved in the regulation of the stability of cell cycle regulator p21(CIP1/WAF1) in human keratinocyte HaCaT cells.
FEBS J. 280, 3840-3853 (2013).