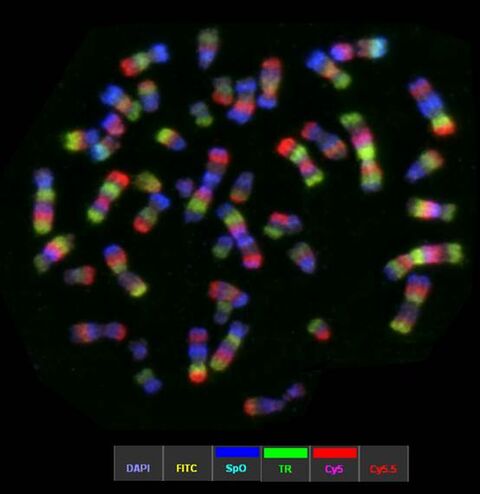
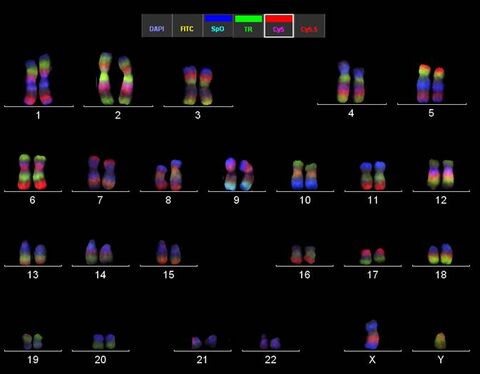
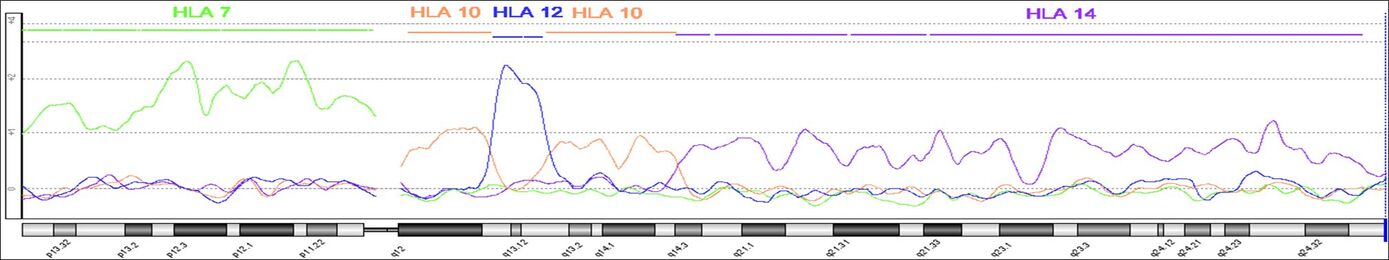
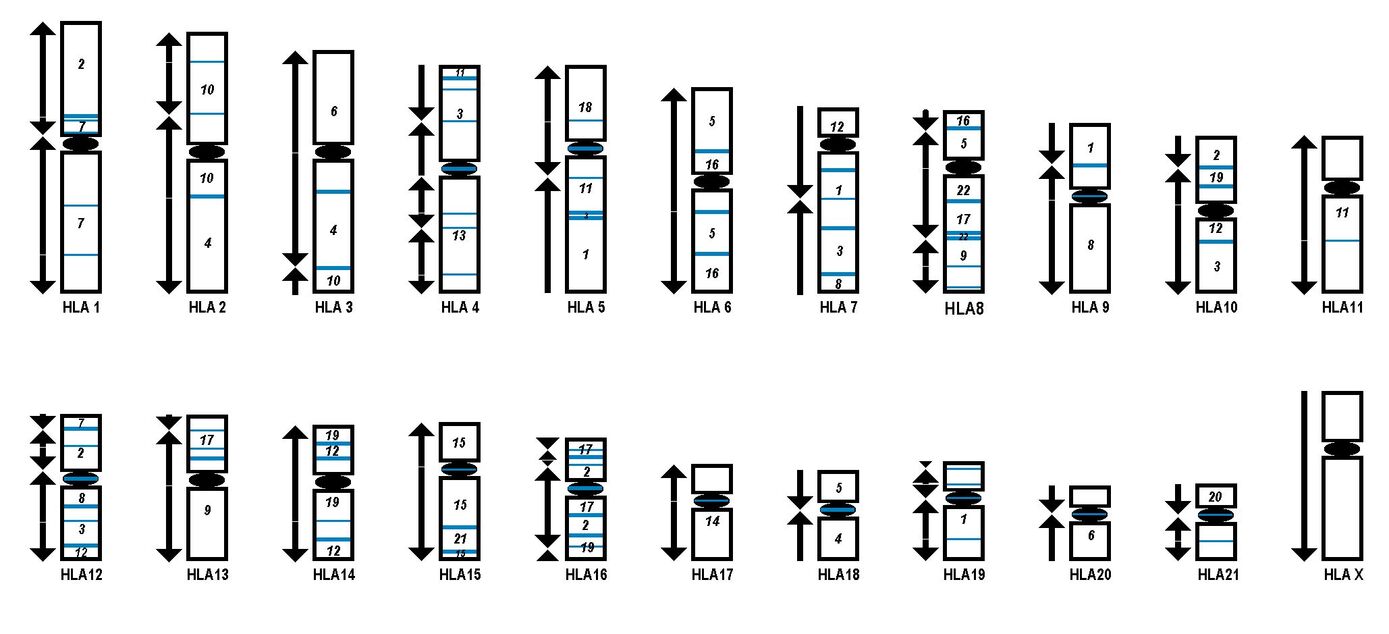
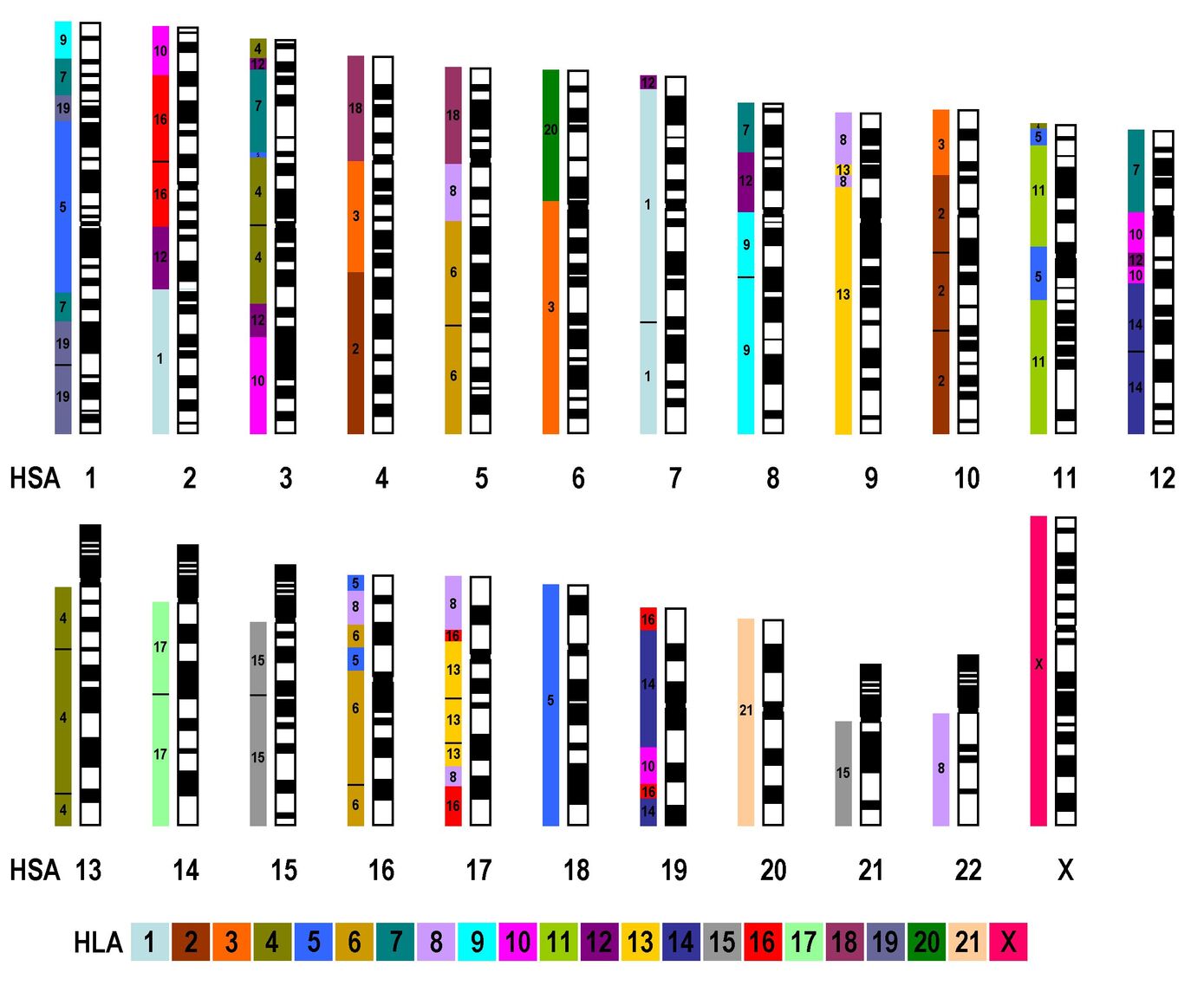
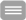mMCB Pseudofarbendarstellung eines Karyogramms eines weiblichen Gorilla beringei beringei. Die hiermit gefundenen Unterschiede zum menschlichen Genom waren in Übereinstimmung mit den Ergebnissen, die mittels Einzel-MCB erzielt worden waren (Mrasek et al., 2001). Die Auswertung des mMCB erfolgt, wie in Weise et al. (2003) erläutert primär an den Fluoreszenz-Profilen und erst in zweiter Linie anhand der hier gezeigten Pseudofarben.
Forschungsschwerpunkte Karyotypevolution
Primaten-FISH
Gesamtgenomische Untersuchungen der Karyotypen der großen Menschenaffen mittels hochauflösendem mMCB und subCTM FISH auf kryptische Veränderungen.
Evolutionäre Bruchpunkte
Eingrenzung und Kartierung evolutionär fixierter Bruchpunkte mittels Mikrosezierung, Reverse-FISH, array-CGH und BACs aus dem Human Genom Projekt.
pod-FISH
Unterscheidung homologer Chromosomen auf Basis von Kopiezahlvarianten (CNV) mittels pod-FISH zur Untersuchung verschiedener Fragestellungen.
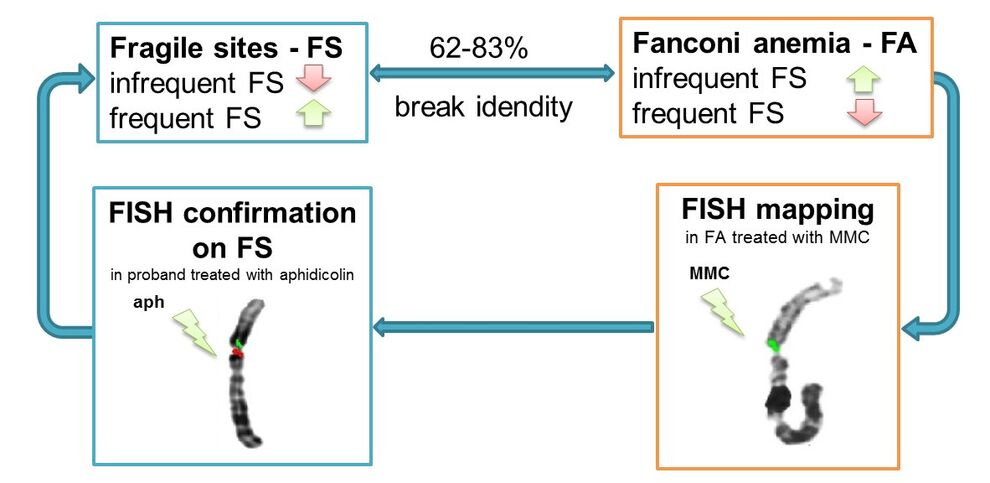
Fragile Sites und Fanconi Anämie
Kartierung von Fragile Sites am Modell der Fanconi Anämie und Abgleich mit Tumorbruchpunkten sowie evolutionär fixierten Bruchpunkten.
Charakterisierung von Zelllinien
Zytogenetische sowie M-FISH, mMCB und MCB basierende Beschreibung von verschiedenen Zelllinien und Spezies.
Karyotypevolution an verschieden behandelten adenomatösen Zelllinien, die zur Karyotyptransformation und Krebsentstehung beitragen.